Dzisiejszy, krótki post będzie dotyczył genu DLGAP2 (zwanego też czasem SAPAP2). Białko kodowane przez ten gen jest ważnym elementem tzw. zagęszczenia postsynaptycznego (ang. post-synaptic density, PSD). Szacuje się, że na zagęszczenie postsynaptyczne składa się z przynajmniej kilkaset białek[1], a niektóre z nich pełnią funkcję rusztowania. Proszę spojrzeć na poniższy obrazek.
Rusztowanie
Uważa się, że wiele z białek wymienionych na powyższym obrazku jest zakonserwowanych genetycznie i nie są charakterystyczne jedynie dla naszego gatunku. Co więcej, przypuszcza się, że zamiany dotyczące tych białek skutkują różnymi zaburzeniami neurologicznymi, w tym ASD. Zaburzenia w funkcjonowaniu synaps są postulowane w autyzmie. Uważa się, że takie białka jak PSD-95, białka z rodziny SAPAP oraz SHANK i jeszcze kilka innych tworzą specyficzne rusztowanie, które organizuje obszar postsynaptyczny i służy jako fundament dla receptorów takich jak NMDAR, AMPAR czy mGluR. Na powyższej ilustracji białko DLGAP2 jest oznaczone jako SAPAP (cała rodzina).
As elegant structures designed for neural communication, synapses are the building bricks of our mental functions. Recently, many studies have pointed out that synaptic protein-associated mutations may lead to dysfunctions of social cognition. Dlgap2, which encodes one of the main components of scaffold proteins in postsynaptic density (PSD), has been addressed as a candidate gene in autism spectrum disorders.
https://openi.nlm.nih.gov/detailedresult.php?img=PMC4113140_2040-2392-5-32-7&req=4
Zaburzenia
O tym w jaki sposób deficyt DLGAP2 może wpływać na zagęszczenie postsynaptyczne mieliśmy okazję wspomnieć przy okazji poprzedniego posta. Pozwolimy sobie ponownie przytoczyć odpowiedni obrazek pokazujący jak wygląda obszar postsynaptyczny u myszek z całkowicie wyłączonym genem DGLAP2. To może nie odpowiadać dokładnie temu co jest widoczne u ludzi z delecją genu DGLGAP2 tylko na jednym chromosomie. Być może w przypadku delecji na jednym chromosomie obserwowana rozbieżność nie jest aż taka wyrazista.
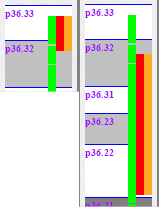
Okazuje się, że zarówno delecje jak i duplikacje w rejonie prążka 8p23 mogą skutkować poważnymi zaburzeniami. I tak, delecja w tym rejonie łączona jest z upośledzeniem umysłowym, autyzmem, mikrocefalią o problemami kardiologicznymi. Z drugiej strony, duplikacja w tym rejonie także wiąże się z ASD i innymi zachowaniami ze spektrum.
8p23 deletions have been associated with mild mental retardation, cardiac abnormalities, microcephaly, and autism (Giglio et al., 2000; Hutchinson, Wilson,&Voullaire, 1992; Tranebjaerg et al., 1984). A duplication at 8p23.2 that intersected and likely disrupted DLGAP2 has been associated with a male with ASD, repetitive behaviors, and moderate receptive/expressive language delay−discussed by both Marshall in 2008 and, after analysis on a higher-resolution array, reported by Pinto and colleagues (Marshall et al., 2008; Pinto et al., 2010).
https://www.ncbi.nlm.nih.gov/pubmed/28407363
Okazuje się, że zaburzenia w genach z rodziny DLGAP/SAPAP widywane są też u osób ze schizofrenią, Zespołem Tauretta czy OCD.
Notably in the present study, we identified several patient-only variants with enhanced promoter activity, suggesting increased DLGAP2 gene expression may contribute to the pathogenesis of schizophrenia.
Our results indicate that DLGAP2 is a susceptible gene of schizophrenia.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3885712/
For example, serial studies supported that the DLGAP3 gene is a promising functional candidate for Tourette syndrome and obsessive compulsive disorder [9], [10], [11], [12]. Pinto et al. discovered the DLGAP2 gene as a novel gene associated with autism spectrum disorders [13]. Recently, our team has identified a private rare mutation (c.1922A>G) of the DLGAP1 gene, which changes lysine to arginine at codon 641 (K641R) in one out of 121 schizophrenia, but not in 275 non-psychotic control subjects [14].
http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0085373
Model mysi
Badań dotyczących genu DLGAP2 jest, niestety, mniej niż by się chciało. Część z nich dotyczy modelu mysiego. Chyba żadna z publikacji nie opisuje potencjalnych interwencji w przypadku delecji genu DLGAP2. Mimo wszystko przyjrzyjmy się temu jakie zachowanie jest widać u myszek z wyłączonym tym genem. Ponieważ badanie dotyczy myszek to trzeba zachowań należytą ostrożność w interpretacji. Okazuje się, że myszki z wyłączonym genem DLGAP2 były przesadnie impulsywne i reagowały nadaktywnością w kontakcie z czymś nowym (zwłaszcza w pierwszych pięciu minutach).
To assess their basic locomotion ability, we used the open-field test (Figure 1E) and found that Dlgap2 −/− mice demonstrated intact locomotion ability since their performances were quite comparable to the WT mice. However, Dlgap2 −/− mice displayed novelty-induced hyperactivity (Figure 1E) in the first 5 minutes of the test specifically, which was likely due to impulsivity [38].
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4113140/
Myszki z wyłączonym genem DLGAP2 były także bardziej agresywne i gorzej się uczyły.
We thus used the resident–intruder task to evaluate the aggressiveness of the Dlgap2 −/− mice. Briefly, mice were single-housed in their home cage for 3 weeks to establish their territory. On the test day, intruders were put in to induce aggressive behaviors in the resident mice. As the results show, Dlgap2 −/− mice displayed an obviously shorter attack latency (Figure 2C). Additionally, the duration and frequency of aggressive behaviors also dramatically increased compared to the WT mice (Figure 2D,E).
We used performance in the water T-maze to determine the repetitive behaviors of Dlgap2 −/− mice. As shown in Figure 3A, there was no significant difference in the number of days to reach the 80% right position criterion between WT and Dlgap2 −/− mice (5.5 and 5.4 days, respectively). All mice reached the criterion on Day 6 and were moved into training session of reversal learning test (Figure 3B). In the learning session of reversal learning test, we found that Dlgap2 −/− mice made a significantly greater number of errors on the first day of learning session in comparison with WT mice (P < 0.05) (Figure 3C). Figure 3D shows the number of errors on each day throughout the 4-day reversal learning session. During reversal learning, both strains improved across days.
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4113140/
Szpilki dendrytyczne
Okazuje się, że anomalie z zagęszczeniem postsynaptycznym to nie jedynie anomalie widoczne po wyłączeniu genu DLGAP2. Kolejna anomalia dotyczy tzw. szpilek dendrytycznych. To co jest widoczne w przypadku delecji DLGAP2 jest przeciwieństwem tego co jest obserwowane w Zespole łamliwego chromosomu X. Proszę spojrzeć na poniższy obrazek. Widać, że gęstość szpilek dendrytycznych jest niższa u myszek z wyłączonym tym genem w porównaniu ze zdrowymi myszkami.
The DLGAP2-encoded SAPAP2 specifically interacts with PSD-95/SAP90 and its related proteins through the guanylate kinase domain. The protein is specifically expressed in neuronal cells, and enriched in the post-synaptic density fraction [8], [16], and plays a pivotal role in the molecular organization of synapses and in neuronal cell signaling. In the case of fragile X syndrome, which is a hereditary form of mental retardation caused by functional absence of Fragile X mental retardation protein (FMRP), Schütt et al. reported that mRNA of SAPAP1, 2 and 3 were part of the targets of the FMRP and increased postsynaptic SAPAP2 levels was observed in the hippocampus of FMRP knock-out mice [29], The increased SAPAP2 level may contribute to the aberrant synaptic function in patients with Fragile X syndrome.
http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0085373
Czy w związku z powyższym, interwencje proponowane w Zespole łamliwego chromosomu X okażą się nieskuteczne u osób z delecją genu DLGAP2? Nie jest to przesądzone. W każdym razie istnieją pewne interwencje modulujące morfologię szpilek dendrytycznych.
Substancja biała
Istnieją pewne poszlaki sugerujące, że delecja genu DLGAP2 może wiązać się z nietypowym rozmiarem substancji białej w mózgu. Zdaje się, że nie ma na to bezpośrednich dowodów, ale anomalie związane z substancją białą widoczne są w przypadku niektórych polimorfizmów tego genu.
Interwencja
Niestety, wydaje się, że delecja genu DLGAP2 jest bardzo rzadka, w związku z czym nie ma finansowej zachęty do pracy nad lekami. Wydaje się, że z uwagi na podobne anomalie występujące zarówno w delecji DLGAP2 jak i delecji SHANK3, pewne interwencje skuteczne dla SHANK3 mogą być także skuteczne dla DLGAP2. Jak jest naprawdę, na ten moment nikt nie wie na pewno. Były już pierwsze próby kliniczne u pacjentów z delecją SHANK3 (https://clinicaltrials.gov/ct2/results?term=Phelan-McDermid&Search=Search) i wspomniano o nich w poprzednim poście. Oprócz wspomnianych tam interwencji być może warto też wspomnieć o podawaniu Rysperydonu, ale w odpowiednio dobranych dawkach.
Another case study examined the effect of risperidone in an 18 year-old woman with PMS. Risperidone was administered daily, and the results showed that 6 mg/day actually worsened behavioral symptoms, including psychomotor agitation, aggression, anxiety, and insomnia, while 1 mg/day of risperidone produced significant improvements using the Clinical Global Impression Improvement Scale [80, 81]. The authors hypothesized that by blocking dopamine 2 receptors, risperidone promotes NMDA transmission and reverses the glutamatergic dysregulation due to loss of SHANK3. Further, the authors argue that the differential effect of high- and low-dose risperidone is consistent with results from animal models, which suggest that risperidone has dose-dependent effects on glutamate receptor subtypes [82].
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4489957/
Konkluzja
Delecje w rejonie prążka 8p23 często skutkują delecją genu DLGAP2. Zebrane dane dotyczące ludzi i badania na modelu zwierzęcym sugerują, że aberacje związane z tym genem mogą mieć znaczący wpływ na funkcjonowanie mózgu i skutkować zaburzeniami ze spektrum autyzmu, schizofrenią, OCD czy Zespołem Tauretta. Wykazano też pewne anomalie związane z zagęszczeniem postsynaptycznym i szpilkami dendrytycznymi. Niestety, na ten moment nie ma w tym przypadku ma zaakceptowanego postępowania medycznego. Istnieją obiecujące próby kliniczne (ale dopiero w pierwszej fazie), które sugerują pewne rozwiązania dla delecji SHANK3. Być może pacjencji z delecją DLGAP2 również pozytywnie zareagują na interwencje stosowane w delecja SHANK3. Czas pokarze (miejmy nadzieję.)
[1] https://en.wikipedia.org/wiki/Postsynaptic_density
[2] https://openi.nlm.nih.gov/detailedresult.php?img=PMC4113140_2040-2392-5-32-7&req=4
[3] https://www.ncbi.nlm.nih.gov/pubmed/28407363
[4] https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3885712/
[5] http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0085373
[6] https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4113140/
[7] https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4552325/
[8] https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4489957/
 http://journal.frontiersin.org/article/10.3389/fncel.2014.00058/full
http://journal.frontiersin.org/article/10.3389/fncel.2014.00058/full https://openi.nlm.nih.gov/detailedresult.php?img=PMC4113140_2040-2392-5-32-7&req=4
https://openi.nlm.nih.gov/detailedresult.php?img=PMC4113140_2040-2392-5-32-7&req=4 https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4113140/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4113140/ https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4552325/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4552325/